Japan
サイト内の現在位置
Jupyter Notebookを使ったFrovedis機械学習 導入編
no.0102021.4.28 データ分析や科学技術計算の分野において、Jupyter NotebookはPythonやRでインタラクティブに試行錯誤しながらプログラミングを行うための環境として幅広く受け入れられています。Pythonのケースで言えば、Jupyter Notebookが内部的にIPythonを呼び出すことで対話的なコードの実行を可能にします。加えて、Jupyter NotebookはMarkdownを使ったドキュメントや画像の埋め込みの他にmatplotlibを使用したグラフを表示するなど、優れたWebベースのコード開発環境を提供します。今回はこのJupyter NotebookとIPythonを使用して、はじめにその設定方法を、続いてSX-Aurora TSUBASAで動作するFrovedisによるデータ処理や機械学習APIの簡単なサンプルコードをデモ形式で御紹介していきます。なお、Frovedisの概要はこちらをご覧ください。
最初にJupyter Notebookを使用するための前準備について。クライアントPC側のWebブラウザからSX-Aurora TSUBASAで動作するJupyter Notebook サーバを使用するための設定、次にFrovedisを使用する際に必要な環境変数設定について説明していきます。
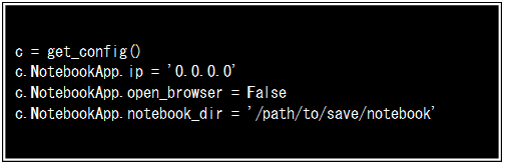
クライアントPCのWebブラウザからJupyter Notebook サーバへ接続可能にするため、サーバ側で~/.jupyter/jupyter_notebook_config.pyにFigure-1に示す設定を追加します 。 c.NotebookApp.ip = ‘0.0.0.0’は、任意のIPアドレスからの接続を許可するために指定しています。c.NotebookApp.open_browserは起動時にブラウザを立ち上げるか選択するためのものですが、ここでは不要(False)を選んでいます。c.NotebookApp.notebook_dirは作業ディレクトリの指定となります。
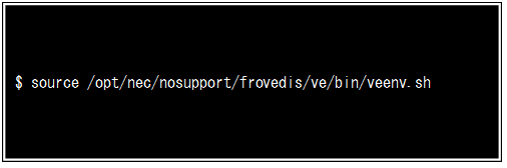
続いて、クライアントPCのターミナルからSX-Aurora TSUBASAにssh接続した後に、Figure-2に示すようにsourceコマンドを使いFrovedis サーバの環境変数を設定します。スクリプトによってPYTHONPATH、LD_LIBRARY_PATHといったPath設定の他、mpirunの実行先をSX-Aurora TSUBASAのベクトルプロセッサに切り替える設定が行われます。なお、sshを通してSX-Aurora TSUBASAにターミナルログインする都度、毎回このスクリプトを実行して環境変数を設定しなおす必要があります。この繰り返しを面倒に感じる方は、変更する環境変数設定を~/.bashrcに記述することで、ターミナルログインの度に行うスクリプト実行の手間を省くことも可能です。
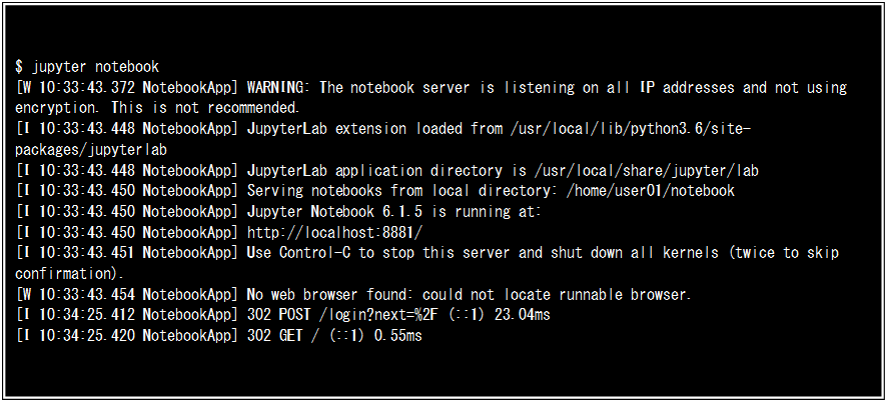
ではSX-Aurora TSUBASAにssh接続したターミナルからJupyter Notebookを起動します。Figure-3のように表示された後に、クライアントPC側のWebブラウザを起動しURL(この場合、http://localhost:8881/login)を入力することでJupyter Notebookの使用準備が整います。では、Jupyter Notebookを使いながらFrovedisによるデータ処理や機械学習APIの簡単なサンプルコードを実行していきましょう。
サンプルコードを実行する前にデータ準備について簡単にふれておきます。Frovedisに限らず機械学習を行う際、多くの場合、最初に学習やテストに使うデータの準備を行います。何らかの方法で収集したデータを使うほか、機械学習のアルゴリズムを試すようなときには、疑似的にデータを作成し学習につかうことも考えられます。クラスタリング学習を例にとると、scikit-learnライブラリの一つであるdatasets.make_blobsを用いた疑似データ作成が思い浮かびます。
こうしたデータの用意はSX-Aurora TSUBASAのホストプロセッサであるXeon(以降、VH)で動作するPythonを使って行う他に、Frovedis サーバに用意されているDistributed matrixやDistributed vectorで行うことも可能です。このFrovedis サーバはSX-Aurora TSUBASAのベクトルプロセッサを使って動作するものであり、VHで動作するPythonとは独立した存在です。必要に応じてFrovedisサーバを呼び出すことで処理をベクトルプロセッサにオフロードします。
Distributed matrixやdistributed vectorを使うメリットは、機械学習する際にSX-Aurora TSUBASAのVHからベクトルプロセッサへのデータ転送する際に発生するコストを削減できる点にあります。VHのPythonで用意したデータをFrovedisサーバで機械学習するにはベクトルプロセッサに転送しなければなりませんが、初めからベクトルプロセッサ側でデータを準備することにより転送することなく学習に落とし込むことができます。加えて、Distributed matrixのデータ格納方法はFrovedisサーバで高速な学習処理が行えるよう配慮がなされています。
Distributed matrixには、データの素性に応じて疎行列を圧縮して格納するCRS matrix (FrovedisCRSMatrix)、密行列を格納する dense matrix (FrovedisRowmajorMatrix, FrovedisColmajorMatrix)、そしてFrovedisBlockcyclicMatrixの3種類が用意されています。 FrovedisCRSMatrix、FrovedisRowmajorMatrix、FrovedisColmajorMatrixは主に機械学習アルゴリズムにデータを送り込む用途で使われます。また、FrovedisBlockcyclicMatrixは格納されたデータを使った演算やScaLAPACKによる連立1次方程式、線形最小二乗問題、固有値問題、特異値問題等を解く用途で用意されています。
CRS matrixは疎行列(行列内の要素に多くの0を含むものです)から0以外のデータだけ抽出して圧縮し格納します。この圧縮により非常に大きなサイズの行列データを効率的にベクトルカードのメモリからベクトルプロセッサに転送して処理することができるようになります。また、最後のBlock Cyclic Matrixは行列をいくつかのブロックに分割し、各ブロックを複数のプロセスとして並列処理して高速化を図るためのものです。
では、Distributed matrixの使い方をJupyter Notebookを使って確認していきます。初めに、FrovedisCRSMatrixを使ったデモンストレーションを行い、続いてBlock Cyclic Matrixによる演算とScaLAPACKを使った線形代数問題解のサンプルを見てみましょう。
import os
import numpy as np
from scipy.sparse import csr_matrix
from frovedis.exrpc.server import FrovedisServer
from frovedis.matrix.crs import FrovedisCRSMatrix
from frovedis.matrix.dense import FrovedisRowmajorMatrix
SciPyの機能を使用して疎行列を作成します。dataに非ゼロのデータを、indicesには非0要素の列情報、indptrに何番目の要素から次の行が始まるか位置情報を記録します。indptrの最後に非ゼロ要素数を追加します。¶
data = np.array([1, 2, 3, 5, 6])
indices = np.array([0, 2, 2, 0, 2])
indptr = np.array([0, 2, 3, 5])
mat = csr_matrix((data, indices, indptr),
dtype=np.float64,
shape=(3, 3))
SciPyで用意した疎行列matの内容を表示します。¶
print(mat)
(0, 0) 1.0 (0, 2) 2.0 (1, 2) 3.0 (2, 0) 5.0 (2, 2) 6.0
row_pointers= mat.indptr
column_index= mat.indices
print(row_pointers, column_index)
[0 2 3 5] [0 2 2 0 2]
一端、疎行列matを密行列形式に変換して行列を表示します。¶
d = csr_matrix.todense(mat)
print(d)
[[1. 0. 2.] [0. 0. 3.] [5. 0. 6.]]
Frovedis serverを起動します。このサンプルでは4つのプロセスを起動して並列処理させます。¶
FrovedisServer.initialize("mpirun -np 4 {}".format(os.environ['FROVEDIS_SERVER']))
fmat = FrovedisCRSMatrix(mat)
Frovedis CRS matrixとNumPy vectorの演算が可能です。¶
vector = np.array([1.0, 2.0, 3.0])
out_ = fmat.dot(np.transpose(vector))
print(out_)
[ 7. 9. 23.]
作成したFrovedis CRS matrixのセーブ、ロードを行います。使用後にfmat、fmat2をメモリから解放します。¶
fmat.save("./result")
fmat.release()
print ("\n-- matrix loaded from text file --")
fmat2 = FrovedisCRSMatrix().load_text("./result")
fmat2.release()
-- matrix loaded from text file --
Frovedis serverを終了します。¶
FrovedisServer.shut_down()
import os
import numpy as np
from frovedis.exrpc.server import FrovedisServer
from frovedis.exrpc.server import *
from frovedis.matrix.dense import *
Frovedis serverを起動します。このサンプルでは4つのプロセスを起動して並列処理させます。¶
FrovedisServer.initialize("mpirun -np 4 {}".format(os.environ['FROVEDIS_SERVER']))
4x4の倍精度浮動小数点データで構成されるnumpy matrixを準備します。¶
mat = np.matrix([[1,2,3,4],[5,6,7,8],[8,7,6,5],[4,3,2,1]],
dtype=np.float64)
先に準備したNumPy行列をFrovedis server側でブロックサイクリック行列fmatに変換します。変換された行列データはベクトルプロセッサのメモリに置かれます。¶
fmat = FrovedisBlockcyclicMatrix(mat)
ブロックサイクリック行列を一端NumPy形式の配列に変換し、表示します。¶
mat2 = fmat.to_numpy_matrix()
print("Frovedis Blockcyclic Matrix => Numpy 2D Matrix", mat2)
Frovedis Blockcyclic Matrix => Numpy 2D Matrix [[1. 2. 3. 4.] [5. 6. 7. 8.] [8. 7. 6. 5.] [4. 3. 2. 1.]]
Frovedis serverで空のRowmajor matrix 'rmat'を作成後、ブロックサイクリック行列同士の演算、転置を行った結果をこの'rmat'に収納します。¶
rmat = FrovedisRowmajorMatrix()
rmat = fmat - fmat
print("m - m: ", rmat.to_numpy_matrix())
rmat = fmat * fmat
print("m * m: ", rmat.to_numpy_matrix())
rmat = ~fmat # fmat.transpose()
print("transpose", rmat.to_numpy_matrix())
m - m: [[0. 0. 0. 0.] [0. 0. 0. 0.] [0. 0. 0. 0.] [0. 0. 0. 0.]] m * m: [[ 51. 47. 43. 39.] [123. 119. 115. 111.] [111. 115. 119. 123.] [ 39. 43. 47. 51.]] transpose [[1. 5. 8. 4.] [2. 6. 7. 3.] [3. 7. 6. 2.] [4. 8. 5. 1.]]
fmat.release()
rmat.release()
ScaLAPACK (Scalable Linear Algebra PACKage) は、分散メモリ型のコンピュータ向けに開発された高性能なルーチン群です。ScaLAPACKは、連立1次方程式、線形最小二乗問題、固有値問題、特異値問題を解くことができます。また、行列の分解や条件数の計算も行うことができます。ScaLAPACKは、PBLAS (Parallel BLAS) と呼ばれる分散メモリ版のレベル1、2、3 BLASを基礎としています¶
ScalaPACKをインポートします。¶
from frovedis.matrix.wrapper import SCALAPACK
3x3倍精度浮動小数点データで構成されるnumpy matrixをはじめに作成します。これをFrovedis server側で循環型分散ブロック行列に変換します。変換された行列データはベクトルプロセッサのメモリに置かれます。¶
mat = np.matrix([[1,1,-1],[-2,0,1],[0,2,1]],
dtype=np.float64)
fmat = FrovedisBlockcyclicMatrix(mat)
LU分解法を用いて'fmat'の逆行列を求めます。はじめにgetrf()を使い'fmat'をLU分解します。¶
res = SCALAPACK.getrf(fmat)
print("getrf info: " + str(res.stat()))
getrf info: 0
続いてLU分解した結果'res'を用いて'fmat'の逆行列を求めます。求めた逆行列は'fmat'に上書きされます。¶
SCALAPACK.getri(fmat,res.ipiv())
print(fmat.to_numpy_matrix())
[[-0.5 -0.75 0.25] [ 0.5 0.25 0.25] [-1. -0.5 0.5 ]]
関数gesv()を用いてAx = bを解きます。¶
fmat = FrovedisBlockcyclicMatrix(mat)
x = np.matrix([[1],[2],[3]], dtype=np.float64)
bcx = FrovedisBlockcyclicMatrix(x)
print("solve Ax = b")
SCALAPACK.gesv(fmat,bcx)
print (bcx.to_numpy_matrix())
solve Ax = b [[-1.25] [ 1.75] [-0.5 ]]
関数gesvd()を用いて'fmat'の特異値分解を行います。¶
bcm = FrovedisBlockcyclicMatrix(fmat)
svd = SCALAPACK.gesvd(bcm)
print("printing the results (umat, svec, vmat)")
(umat,svec,vmat) = svd.to_numpy_results()
print (umat)
print (svec)
print (vmat)
printing the results (umat, svec, vmat) [[-7.07106781e-01 7.07106781e-01 2.22044605e-16] [-7.07106781e-01 -7.07106781e-01 -3.33066907e-16] [-1.38492816e-16 -3.58163096e-16 1.00000000e+00]] [2.44948974 2. 1.22474487] [[ 5.77350269e-01 -5.77350269e-01 -5.77350269e-01] [-7.07106781e-01 -7.07106781e-01 1.03443839e-16] [-4.08248290e-01 4.08248290e-01 -8.16496581e-01]]
使用後にデータのメモリー解放とFrovedis server停止を行います。¶
fmat.release()
res.release()
bcm.release()
FrovedisServer.shut_down()
ここまではデータの取り扱いでしたが、次にFrovedis サーバでLogistic RegressionとPCAによる機械学習を行います。使用するデータはscikit-learn からbreast cancerデータセットをロードします。初めに教師あり学習のLogistic Regressionを使いデータの分類を行い、次にPCAによる次元圧縮を利用した2次元でのデータ可視化を実施します。
scikit-learn breast cancerrデータセットを用いたFrovedis版LogisticRegressionとPCAの使用例¶
import os
import numpy as np
import pandas as pd
from sklearn.datasets import load_breast_cancer
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
import seaborn as sns
Frovedis serverを使用するためFrovedisServerとLogisticRegression、PCAをインポートします。¶
from frovedis.exrpc.server import FrovedisServer
from frovedis.mllib.linear_model import LogisticRegression
from frovedis.decomposition import PCA
scikit-Learnのbreast cancerデータセットをロードします。¶
X, y = load_breast_cancer(return_X_y=True)
frovedis_serverを起動します。¶
FrovedisServer.initialize("mpirun -np 4 {}".format(os.environ['FROVEDIS_SERVER']))
C = 100
max_iter=10000
solver = "sag"
LogisticRegressionにおけるテストスコアを算出します。¶
clf = LogisticRegression(random_state=0, solver=solver, C=C, max_iter=max_iter).fit(X, y)
y_pred = clf.predict(X)
score = 1.0 * sum(y == y_pred) / len(y)
print("score: {}".format(score))
score: 0.9226713532513181
PCAによるbreast cancerデータの可視化¶
PCA適用前にデータをスケール変換し分散が1の特徴量にします。¶
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
breast cancerデータにPCAモデルを適用し、最初の2つの主成分に対してデータポイントを変換します。¶
pca = PCA(n_components=2)
pca.fit(X_scaled)
X_pca = pca.transform(X_scaled)
2Dで2つの主成分をグラフ化します。¶
plt.figure(figsize=(10,7))
sns.scatterplot(x=X_pca[:,0], y=X_pca[:,1], hue=y, palette=['yellow', 'green'])
plt.title("2D scatter plot")
plt.xlabel("PC1")
plt.ylabel("PC2")
Text(0, 0.5, 'PC2')
FrovedisServer.shut_down()
以上、Jupyter Notebookを使いSX-Aurora TSUBASAのFrovedis サーバに接続するための前準備と、Distributed matrixのデモ、そしてFrovedis サーバが提供する機械学習アルゴリズムを使った簡単なサンプルコードをご覧いただきました。
関連リンク
 AL/ML開発向けオープンソースプラットフォーム「Frovedis」
AL/ML開発向けオープンソースプラットフォーム「Frovedis」
 SX-Aurora TSUBASAテクニカルセミナー特設サイト
SX-Aurora TSUBASAテクニカルセミナー特設サイト
本コラム内容を踏まえたテクニカルセミナー動画公開中。第1回講演3参照。